pyMC5の記事の5記事目です。
今回はpyMCをつかって変更点の検出をやってみます。といっても、題材が変更点の検出というだけでメインで取り上げたいのはデータ列の途中で期待値が変わるモデルを扱いたいってのと、事後分布からのサンプリングをやりたいっていう点の2点です。
題材の紹介
題材としては、少し古い本なのですが「Pythonで体験するベイズ推論」という、キャメロン・デビッドソン=ピロンさんの本から持ってきます。これの、受信するメッセージ数の変化がテーマです。
なぜこんな古い本を題材にしたかというと、新しい本を題材にするとそのままコードを書き写す感じになるので勉強にならないからです。この本のコードはpyMC3なのでpyMC5に焼き直すとコードは大幅に変わります。
サンプルデータはこちらのリポジトリにあります。
ただ、ここからダウンロドしてくるのも手間ですし、ただの整数列なのでそのままこの記事にも書いておきます。ちなみに、全部で74日分です。
data = [13, 24, 8, 24, 7, 35, 14, 11, 15, 11, 22, 22, 11,
57, 11, 19, 29, 6, 19, 12, 22, 12, 18, 72, 32, 9, 7, 13,
19, 23, 27, 20, 6, 17, 13, 10, 14, 6, 16, 15, 7, 2, 15,
15, 19, 70, 49, 7, 53, 22, 21, 31, 19, 11, 18, 20, 12, 35,
17, 23, 17, 4, 2, 31, 30, 13, 27, 0, 39, 37, 5, 14, 13, 22
]結果は最後に図示しますが、これ途中から件数が増えているように見えるのですよ。
それが、何日目から増えていて、その前後は期待値何件だったのか、というのをpyMCで推論していきます。
pyMCによるモデリング
ではやっていきましょう。
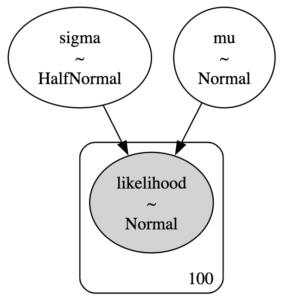
データが整数値なので、メッセージの件数はポアソン分布に従うとし、前半と後半で期待値$\lambda$が違うものとします。それぞれの$\lambda$はガンマ分布から持ってきましょう。
そして、変化する日はtauという変数名で離散一様分布からサンプリングします。
tauより前の日と以降の日で期待値が変わる点については、pm.math.switchを使って実装しました。この辺は本のコードと違います。
import matplotlib.pyplot as plt
import pymc as pm
import arviz as az
model = pm.Model()
with model:
# 変化前の期待値
lambda_1 = pm.Gamma('lambda_1', alpha=2, beta=1)
# 変化後の期待値
lambda_2 = pm.Gamma('lambda_2', alpha=2, beta=1)
# 変化日
tau = pm.DiscreteUniform('tau', lower=0, upper=len(data)-1)
# idxは各データ点のインデックス
idx = np.arange(len(data))
# lambda_の値がtauの値によって決定される
lambda_ = pm.math.switch(tau >= idx, lambda_1, lambda_2)
# データの尤度
obs = pm.Poisson('obs', mu=lambda_, observed=data)
# サンプリング
trace = pm.sample(draws=10000, tune=10000, chains=2)サンプリングは少し多めに行っています。
結果を見ておきましょう。
print(az.summary(trace))
"""
mean sd hdi_3% hdi_97% mcse_mean mcse_sd ess_bulk ess_tail r_hat
tau 43.221 0.875 42.000 44.000 0.016 0.011 3054.0 4512.0 1.0
lambda_1 17.406 0.617 16.275 18.576 0.004 0.003 18857.0 14591.0 1.0
lambda_2 22.035 0.859 20.354 23.597 0.007 0.005 15301.0 14084.0 1.0
"""
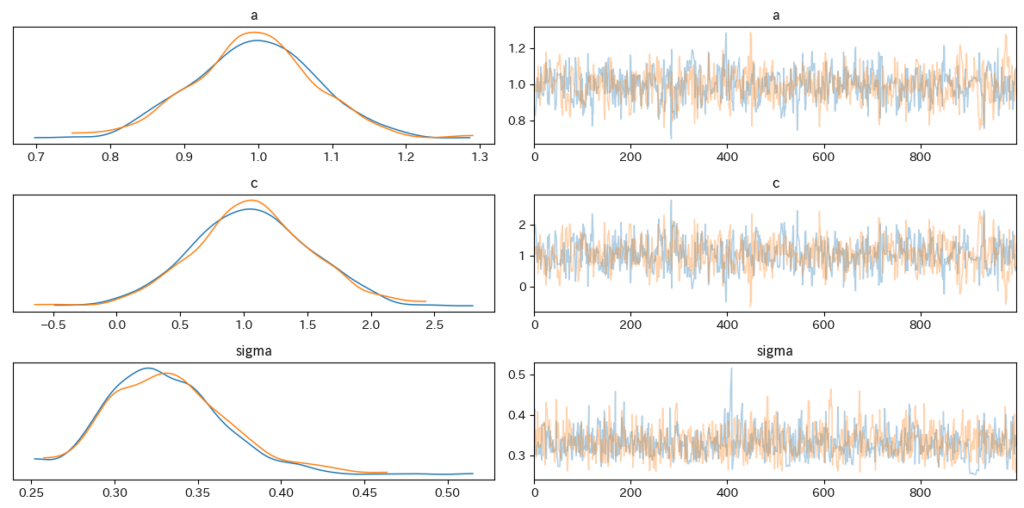
az.plot_trace(trace)
plt.tight_layout()トレースを可視化したものがこちらです。
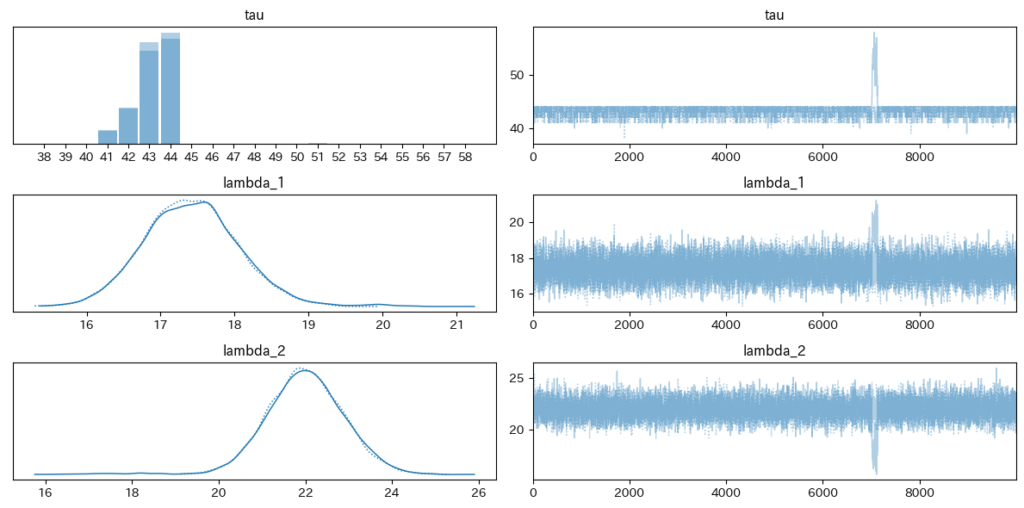
一瞬怪しいところがありますがtau = 44 あたりで変更しているのがわかりますね。
そこを閾にlambdaが増えていそうです。(17.4くらいから22くらいに。)
0日目から40日目くらいはでは、期待値17.4くらいで、45日目以降は22くらいと見て良さそうです。さて、その途中の41〜44日目はどう考えましょう?となった時に便利なのが事後分布からのサンプリングです。tauの分布を考慮していちいち計算しなくても、さっとサンプリングしてしまうことでそれぞれの日のメッセージ受信数の期待値の概算がわかります。
事後分布からのサンプリングに使うのが、 pm.sample_posterior_predictive です。これはモデルではなく、traceの方を渡してサンプリングを行います。サンプリングされるサイズが、最初に推論した時のステップ数に依存してしまうので、実はさっきサンプリングする時にdrawを大きめの値にしていたのです。draws=10000, chains=2 だったので、20000サンプルが得られます。
参考: pymc.sample_posterior_predictive — PyMC dev documentation
さて、やってみます。
# 既存のモデルとトレースを使用して、事後予測サンプルを生成
with model:
# traceから事後予測サンプルを生成する際の修正
posterior_predictive = pm.sample_posterior_predictive(trace, var_names=['obs'])
# 'obs'キーを使用して事後予測サンプルのデータを取得
posterior_obs = posterior_predictive.posterior_predictive['obs'].values
# 事後予測サンプルの期待値(平均)を計算
expected_values = np.mean(posterior_obs, axis=(0, 1))axis=(0, 1) としているのは元の posterior_obs.shape が (2, 10000, 74) だからです。
これで、obsのサンプリング結果の期待値が得られました。元のデータと合わせて可視化してみましょう。
fig = plt.figure(facecolor="w")
ax = fig.add_subplot(1, 1, 1)
ax.bar(range(len(data)),data)
ax.plot(expected_values, c="orange")
plt.show()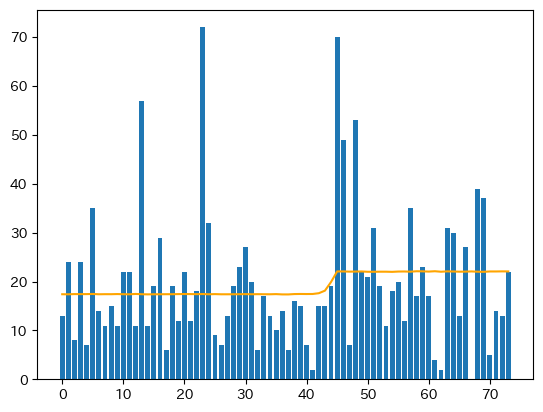
いい感じですね。