pyMCの3つ目の記事です。
このブログではまだ超単純なものしか扱っていませんが、pyMCではかなり柔軟にモデルを構築できます。そうなってくると、自分が実装したモデルが妥当なものなのか、思うような事象を表現できているのかといったことをサンプリング前に確認しておきたくなるものだと思います。
そのような場合に備えて、pyMCでは、ベイズ推論を行う前に事前分布からそのままサンプリングを行い結果を確認するメソッドとして、 pm.sample_prior_predictive() というものが用意されています。
参考: pymc.sample_prior_predictive — PyMC dev documentation
使い方は簡単で、モデルを組んだ後にsample()の代わりにこれを呼び出すだけです。引数としてはサンプリングしたいデータ数を渡します。
では具体的にやってみましょう。今回は二項分布あたりを扱ってみましょうかね。$n=10$くらいで、$p$は0から1の一様分布からサンプリングしてみましょう。通常二項分布って$np$を期待値として山形になるのですが、$p$が固定されていないのでそうはならない例をお見せできると思います。
import pymc as pm
import arviz as az
import matplotlib.pyplot as plt
with pm.Model() as model:
# 事前分布を設定
p = pm.Uniform('p', lower=0, upper=1)
y = pm.Binomial('y', n=10, p=p)
# 事前分布からサンプリング
prior_predictive = pm.sample_prior_predictive(samples=1000)このサンプリングはかなり短時間で終わります。複雑なモデルの通常のサンプリングは結構時間がかかるので、その意味でも事前確認をしておくメリットはありますね。
サンプリングしたら結果を確認します。prior_predictive って変数に結果が確認されていますが、`prior_predictive.prior[“p”]`や`prior_predictive.prior[“y”]`のような形式でサンプリング結果を取り出せます。
arviz使って確認しましょう。
fig = plt.figure(figsize=(12, 5), facecolor="w")
ax = fig.add_subplot(1, 2, 1, title="p")
az.plot_dist(prior_predictive.prior["p"], ax=ax)
ax = fig.add_subplot(1, 2, 2, title="y")
az.plot_dist(prior_predictive.prior["y"], ax=ax)
plt.show()結果がこちらです。
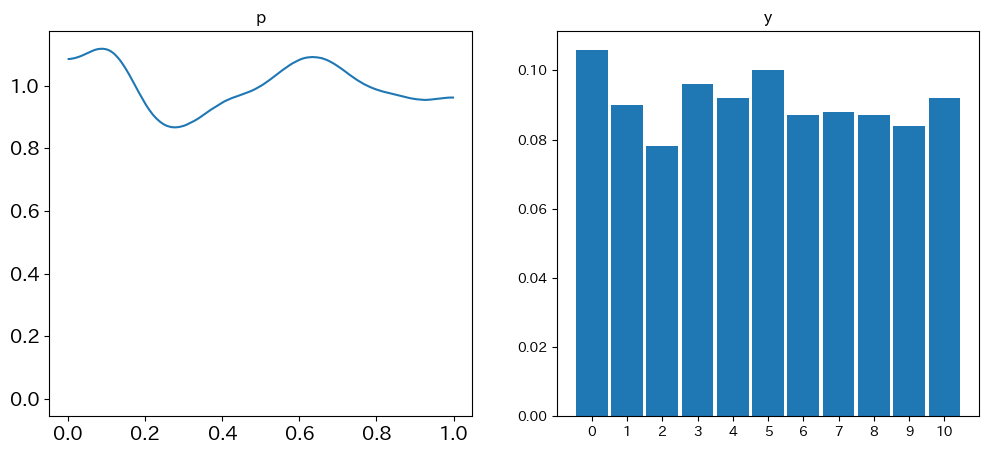
サンプルサイズ(1000と設定しました。デフォルトは500です。)が小さいのか、ちょっとpの分布がボコボコしていますが概ね想定通りの結果が得られましたね。