NetworkXでグラフを可視化する時、
2次元だとエッジが多すぎていわゆる毛玉状態になり、わけがわからないけど3次元だと少しマシになるということがあったので、3次元でプロットする方法を紹介しておきます。
公式ドキュメントの 3D Drawing のページを見ると、
Mayavi2 というのを使う方法が紹介されています。
ただ、僕がこれを使ったことがないのと、Matplotlibで十分できそうだったので、Matplotlibでやってみました。
Mayavi2 はこれはこれで便利そうですし、可視化の幅を広げられそうなので近いうちに試します。
まず、可視化するグラフデータを生成します。
今回はいつもみたいにランダム生成ではなく、エッジを具体的に指定して構築しました。
出来上がるのはサッカーボール型の多面体です。
(実はこのデータ生成の方が3次元プロットより苦労しました。)
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
import networkx as nx
import numpy as np
# エッジデータを生成
edge_list = [
(0, 1), (1, 2), (2, 3), (3, 4), (4, 0),
(0, 5), (1, 6), (2, 7), (3, 8), (4, 9),
(5, 10), (10, 11), (11, 6), (6, 12), (12, 13),
(13, 7), (7, 14), (14, 15), (15, 8), (8, 16),
(16, 17), (17, 9), (9, 18), (18, 19), (19, 5),
(11, 20), (20, 21), (21, 12), (13, 22), (22, 23),
(23, 14), (15, 24), (24, 25), (25, 16), (17, 26),
(26, 27), (27, 18), (19, 28), (28, 29), (29, 10),
(21, 30), (30, 31), (31, 22), (23, 32), (32, 33),
(33, 24), (25, 34), (34, 35), (35, 26), (27, 36),
(36, 37), (37, 28), (29, 38), (38, 39), (39, 20),
(31, 40), (40, 41), (41, 32), (33, 42), (42, 43),
(43, 34), (35, 44), (44, 45), (45, 36), (37, 46),
(46, 47), (47, 38), (39, 48), (48, 49), (49, 30),
(41, 50), (50, 42), (43, 51), (51, 44), (45, 52),
(52, 46), (47, 53), (53, 48), (49, 54), (54, 40),
(50, 55), (51, 56), (52, 57), (53, 58), (54, 59),
(55, 56), (56, 57), (57, 58), (58, 59), (59, 55),
]
# 生成したエッジデータからグラフ作成
G = nx.Graph()
G.add_edges_from(edge_list)
さて、データができたので早速3次元空間にプロットしてみましょう。
方法は簡単で、以前紹介したmatplotlibの3次元プロットの方法で、
ノードとエッジを順番に出力するだけです。
ノードの方はこちらの記事が参考になります。
参考: matplotlibで3D散布図
エッジの方はまだ直接的に消化はしていませんが、2次元空間に直線を引く時と同様に、
ax.plot で描けます。
実際にやってみたのが以下のコードです。
比較用に2次元にプロットしたものを横に並べました。
# spring_layout アルゴリズムで、3次元の座標を生成する
pos = nx.spring_layout(G, dim=3)
# 辞書型から配列型に変換
pos_ary = np.array([pos[n] for n in G])
# ここから可視化
fig = plt.figure(figsize=(20, 10), facecolor="w")
ax = fig.add_subplot(121, projection="3d")
# 各ノードの位置に点を打つ
ax.scatter(
pos_ary[:, 0],
pos_ary[:, 1],
pos_ary[:, 2],
s=200,
)
# ノードにラベルを表示する
for n in G.nodes:
ax.text(*pos[n], n)
# エッジの表示
for e in G.edges:
node0_pos = pos[e[0]]
node1_pos = pos[e[1]]
xx = [node0_pos[0], node1_pos[0]]
yy = [node0_pos[1], node1_pos[1]]
zz = [node0_pos[2], node1_pos[2]]
ax.plot(xx, yy, zz, c="#aaaaaa")
# 比較用 : 通常の2次元軸へのプロット
ax = fig.add_subplot(122)
nx.draw_networkx(G, edge_color="#aaaaaa")
# 出来上がった図を表示
plt.show()
このコードで以下の図が出力されました。
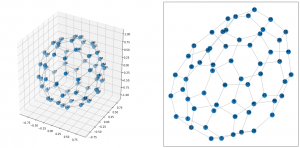
3次元の方がサッカーボール として綺麗な形になっているのがみて取れると思います。
座標軸の数値はいらないのでこれを消すなどの工夫を加えたらもっと良いかもしれませんね。
blog記事には静止画で貼り付けましたが、
jupyter notebook で実行する時は、
%matplotlib notebook を実行しておくと、
3次元プロットはグリグリ動かして確認ができます。
結構便利なので機会があれば試してみてください。
追記(2022/11/16) : エッジの描写についての解説
3次元にプロットするところの、エッジ(線)の描写部分について質問いただきましたので、解説します。
該当コードはここですね。
for e in G.edges:
print(e)
node0_pos = pos[e[0]]
node1_pos = pos[e[1]]
xx = [node0_pos[0], node1_pos[0]]
yy = [node0_pos[1], node1_pos[1]]
zz = [node0_pos[2], node1_pos[2]]
ax.plot(xx, yy, zz, c="#aaaaaa")
まず、最初のfor文ですが、これはグラフのエッジをループさせています。変数eエッジの中の一つが格納され、それがどのノードからどのノードへのエッジなのかの情報がただのタプルとして入ってます。1個目だけprintしてみます。一番最初に作ったエッジデータの1個目ですね。
for e in G.edges:
print(type(e))
print(e)
break # 打ち切り
"""
<class 'tuple'>
(0, 1)
"""
エッジが(0, 1)ですから、まずノード0からノード1へ線をひこう、というのが以降の処理です。そのために、ノード0とノード1はどの座標に配置されているのかの情報が必要になります。
その座標が spring_layout ってアルゴリズムで推定して、ノード:座標の形でposって変数に辞書で入ってます。(上の方のコード参照)
中身を見ておきましょう。全ノード分含まれているのですが、最初の5件blogに載せます。
from pprint import pprint
pprint(pos)
"""
{0: array([-0.6114604 , 0.62195763, 0.31867187]),
1: array([-0.5150625 , 0.74024425, -0.01842863]),
2: array([-0.17471886, 0.96503904, 0.00644528]),
3: array([-0.02757867, 0.98970947, 0.35055788]),
4: array([-0.30497656, 0.77198962, 0.53307487]),
# 以下略
"""
e = (0, 1) ですから、 e[0] = 0, e[1] = 1です。(最初のedgeはインデックスと中身が一致しててややこしく、すみません)
これを使って、エッジが繋いでる2頂点の座標を取得します。アルゴリズムが乱数使っているので具体的な値は実行するたびに変わりますのでご注意ください。
node0_pos = pos[e[0]]
node1_pos = pos[e[1]]
# 一つ目のノードの座標
print(node0_pos)
# [-0.6114604 0.62195763 0.31867187]
# 二つ目のノードの座標
print(node1_pos)
# [-0.5150625 0.74024425 -0.01842863]
具体的な座標が定まったので、この2点の間に線を引きます。これは、Axes3Dをimportした状態のmatplotlibのplotメソッドで実行します。
この時にax.plot(1点目の座標, 2点目の座標)と渡すと動かないのです。
2次元のplotにおいても ax.plot(x座標の一覧, y座標の一覧) とデータを渡すように、3次元plotでもax.plot(x座標の一覧, y座標の一覧, z座標の一覧)とデータを渡す必要があります。
xxとかyyとか変な変数名で恐縮ですが、それを続くコードでやってます。
# x座標, y座標, z座標をそれぞれ取り出し
xx = [node0_pos[0], node1_pos[0]]
yy = [node0_pos[1], node1_pos[1]]
zz = [node0_pos[2], node1_pos[2]]
# 中身確認
print(xx)
# [-0.6114604015979618, -0.5150625045997115]
print(yy)
# [0.6219576319265612, 0.740244253223188]
print(zz)
# [0.3186718713992598, -0.01842863446943393]
そして、出来上がったxx,yy,zz を ax.plot()に渡してエッジが1本引けたことになります。
c=”#aaaaaa” はただの色設定(灰色)なので問題ないと思います。
これで1つ引けるので、あとはfor文で各エッジを変数eに格納して順次繰り返しています。