前回の記事の続きです。
参考:scipyで階層的クラスタリング
前回の記事で階層的クラスタリングを実行し可視化するところまで紹介しましたが、
今回は一歩戻ってlinkage関数の戻り値の中身を見てみます。
とりあえず、 linkage matrix をprintして結果を見てみましょう。
from sklearn.datasets import load_iris
from scipy.cluster.hierarchy import linkage
X = load_iris().data[::10, 2:4]
print(X.shape) # (15, 2)
# ユークリッド距離とウォード法を使用してクラスタリング
z = linkage(X, metric='euclidean', method='ward')
print(z.shape) # (14, 4)
print(z)
# 以下出力
[[ 2. 3. 0.1 2. ]
[ 0. 1. 0.1 2. ]
[12. 14. 0.14142136 2. ]
[ 4. 16. 0.2081666 3. ]
[ 6. 8. 0.31622777 2. ]
[ 5. 9. 0.36055513 2. ]
[ 7. 11. 0.36055513 2. ]
[15. 18. 0.39072582 5. ]
[10. 17. 0.43969687 3. ]
[13. 23. 0.73598007 4. ]
[20. 21. 1.0198039 4. ]
[19. 25. 2.00831604 6. ]
[24. 26. 3.72312593 10. ]
[22. 27. 9.80221064 15. ]]
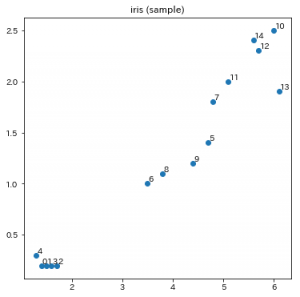
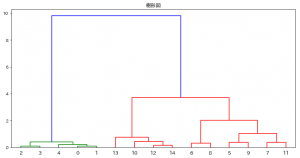
前回の記事で可視化したのと同じデータなので、以降の説明は前回の記事中の図と見比べながら読むとわかりやすいと思います。
結果のlinkage matrixは、z.shape の値から分かる通り、14行4列の行列の形をしています。
で、この14という値は、元のデータの個数15個から1減らした値です。
階層的クラスタリングのプロセスの中で、1個ずつグルーピングして集約し、もともと15個あったグループを1つにまとめるのでこうなってます。
そして、列ですが、pythonのインデックスいうと 0列目と1列目はあたらに同じグループに含まれるデータのインデックス、
2列目はそれらの要素orクラスタ間の距離、3列めはそこで新たに作られたクラスタに含まれれるデータの個数を意味します。
具体的には、次のデータは、X[2]とX[3]の距離が0.1で、この二つをまとめて要素が2個のクラスタを作ったことを意味します。
[ 2. 3. 0.1 2. ]
そして、明示はされていませんが、その新しいクラスタには、インデックス15が振られます。(元のデータが0~14の15個なので。)
同様に、次のデータで0と1がまとめられてインデックス16のクラスタが作られます。
[ 0. 1. 0.1 2. ]
で、このインデックス16のクラスタは次のデータで4番目の要素とグルーピングされて、要素数3個のクラスタになります。
[ 4. 16. 0.2081666 3. ]
前回の記事のデンドログラムで確かに0と1でできたクラスタに4が合流しているのが描かれていますね。
このようにして、 linkage matrix の中身を直接読むことができます。