昨日の記事が移動平均だったので、今日は指数平滑移動平均を扱います。
初めて知った日は衝撃だったのですが、pandasには指数平滑移動平均を計算する専用の関数が用意されています。
(pythonを使い始める前はExcel VBAでいちいち実装していたので非常にありがたいです。)
馴染みがない人もいると思いますので軽く紹介しておきます。
元のデータを${x_t}$とし、期間$n$に対して指数平滑移動平均${EWMA_t}$は次のように算出されます。
$$
\begin{align}\alpha &= \frac{2}{1+n}\\
EWMA_0 &= x_0\\
EWMA_t &= (1-\alpha)*EWMA_{t-1} + \alpha * x_t
\end{align}
$$
3番目の式を自分自身に逐次的に代入するとわかるのですが、
$EWMA_t$は、$x_t$から次のように算出されます。
$$
EWMA_t = \alpha\sum_{k=0}^{\infty}(1-\alpha)^k x_{t-k}
$$
$(1-\alpha)$の絶対値は1より小さいので、この無限級数の後ろの方の項は無視できるほど小さくなります。
結果的に、過程${x_t}$の最近の値に重みを置いた加重平均と見做せます。
さて、早速ですが計算してみましょう。
pandasのDataFrameおよび、Seriesに定義されているewm関数を使います。
pandas.DataFrame.ewm
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
# データ作成
data = pd.Series(np.random.normal(0, 100, 200).cumsum() + 20000)
# 指数平滑移動平均の計算
data_ewm = data.ewm(span=10).mean()
# 可視化
plt.rcParams["font.size"] = 14
fig = plt.figure(figsize=(12, 7))
ax = fig.add_subplot(1, 1, 1)
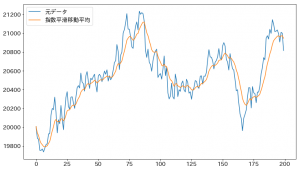
ax.plot(data, label="元データ")
ax.plot(data_ewm, label="指数平滑移動平均")
plt.legend()
plt.show()
出力がこちら。
ここで一つ注意する点があります。
data_ewm = data.ewm(span=10).mean()
という風に、spanという変数名で期間$10$を渡しています。
ドキュメントを読んでいただくとわかるのですが、span=をつけないと、
comという別の変数に値が渡され、$\alpha$の計算が、
$\alpha=1/(1+com)$となり、結果が変わります。
また、spanやcomを使う以外にも、alpha=で$\alpha$のあたいを直接指定することも可能です。