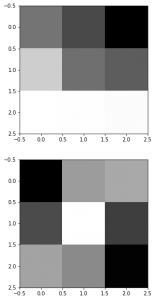この前の記事で、scikit-learnのニュース記事のジャンルをロジスティック回帰で予測するというモデルを作ってみました。
参考:scikit-learnのニュースデータをロジスティック回帰で分類
今回はアプローチを変えて、トピックモデルを試してみようと思います。
どちらかというと、20newsのデータセットでもう少し何かやりたいというのが主目的で、
トピックモデルの理論的な説明については今回は省略します。
興味のあるかたへは、講談社から出ている岩田具治先生の、 トピックモデル (機械学習プロフェッショナルシリーズ)
が非常にわかりやすかったのでおすすめです。ページ数も少なめでありがたい。
(数式が多くて書くのが大変なのですがゆくゆくは時系列分析みたいにこのブログでも説明したい。)
さて、pythonでトピックモデルを実装するには gensim を使うのが一般的のようです。
gensim topic modelling for humans
ただ、今回はいつも使っているscikit-learnでやってみました。
(gensimはword2vec等で使ってるのですがscikit-learnに比べると少し苦手。)
scikit-learnでトピックモデルを実装するために読むドキュメントはこちら。
sklearn.decomposition.LatentDirichletAllocation
Topic extraction with Non-negative Matrix Factorization and Latent Dirichlet Allocation
サンプルコードと同じことをしてもしょうがないので、少し工夫をしています。
・サンプルデータのカテゴリーを前回の記事同様に5個に絞る(その代わりそのカテゴリの全データを使用)
・カテゴリーごとに各文章のトピッックを可視化
前置きが長くなりましたが、やってみましょう。
必要ライブラリーのインポートとデータの読み込み
from sklearn.feature_extraction.text import CountVectorizer
from sklearn.decomposition import LatentDirichletAllocation
from sklearn.datasets import fetch_20newsgroups
import matplotlib.pyplot as plt
import numpy as np
remove = ('headers', 'footers', 'quotes')
categorys = [
"rec.sport.hockey",
"soc.religion.christian",
"sci.med",
"comp.windows.x",
"talk.politics.mideast",
]
twenty_news = fetch_20newsgroups(
subset='all',
remove=remove,
categories=categorys
)
X = twenty_news.data
続いて、単語の出現頻度を数え、LDAのモデルを構築して学習します。
トピック数は カテゴリー数と同じ5でも試したのですが、
どうやら6か7にして、あまり重要でない単語を引き受けるトピックを作った方が納得性の高いものになりました。
サンプルコードは20カテゴリーを10トピックでうまく処理できているのに何故だろう?
# 単語の出現頻度データを作成
tf_vectorizer = CountVectorizer(max_df=0.90, min_df=5, stop_words='english')
tf = tf_vectorizer.fit_transform(X)
len(tf_vectorizer.get_feature_names())
# LDAのモデル作成と学習
lda = LatentDirichletAllocation(
n_components=7,
learning_method='online',
max_iter=20
)
lda.fit(tf)
それでは、学習した7個のトピックについて、それぞれの頻出語をみてみます。
features = tf_vectorizer.get_feature_names()
for tn in range(7):
print("topic #"+str(tn))
row = lda.components_[tn]
words = ', '.join([features[i] for i in row.argsort()[:-20-1:-1]])
print(words, "\n")
出力は下記の通りです。(乱数の影響で、モデルの学習をやり直すと結果は変わります。)
topic #0
god, people, think, don, know, just, like, does, say, believe, jesus, church, time, way, did, christ, things, good, christian, question
topic #1
25, 10, 11, 12, 14, 16, 15, 17, 20, 13, 18, 19, 55, 30, la, period, 24, 21, pit, 92
topic #2
armenian, armenians, turkish, people, turkey, armenia, turks, greek, genocide, russian, azerbaijan, government, history, muslim, university, soviet, war, 000, ottoman, killed
topic #3
game, don, said, team, just, didn, hockey, like, know, went, year, time, games, think, got, people, going, did, ll, came
topic #4
israel, jews, jewish, israeli, arab, state, people, world, right, public, arabs, rights, human, war, anti, peace, adl, states, country, palestinian
topic #5
medical, health, disease, cancer, patients, use, new, hiv, doctor, season, good, treatment, years, aids, high, drug, number, time, information, vitamin
topic #6
edu, use, file, window, com, server, program, dos, windows, available, motif, using, version, widget, sun, set, display, mit, x11, information
#1があまり意味のない数値を引き受けてくれていますが、
それ以外は、トピックごとに、宗教や国際的な話題、スポーツに医療に、コンピューターなどの単語が分類されています。
最後に、元の各テキストが、カテゴリーごとに妥当なトピック(話題)を持つと判定さているのか可視化してみてみましょう。
どんな可視化方法が一番わかりやすいか色々試したのですが、カラーマップが比較的良さそうでしたので紹介します。
(このほか箱ひげ図などもそこそこ綺麗に特徴が出ましたが。)
topic_data = lda.transform(tf)
fig = plt.figure(figsize=(6, 25))
for i in range(5):
ax = fig.add_subplot(6, 1, 1+i)
im = ax.pcolor(topic_data[twenty_news.target == i], vmax=1, vmin=0)
fig.colorbar(im)
# 軸の設定
ax.set_xticks(np.arange(7) + 0.5, minor=False)
ax.set_xticklabels(np.arange(7))
ax.set_title(twenty_news.target_names[i])
plt.show()
出力がこちら。

概ね、カテゴリーごとに別のトピックに分類されているのがみて取れます。