前回の記事で紹介したJupyter Notebookのウィジェット(ボタン)の簡単な応用事例です。
参考: Jupyter Notebook でボタンを使う
この記事でいくつか事例を紹介しようと思いますが、まずは機械学習等でテキストデータの分類モデルをつくつる時の教師データ作成(アノテーション)のツールを作ってみようと思います。
何かしらテキストデータを受け取って、事前に定義されたいくつかのカテゴリ(ラベル)の中から1個選んで、「これはカテゴリ0だ」とか「こいつはカテゴリ3だ」みたいなのを記録してくツールですね。
とりあえず、使うデータの準備と必要なライブラリを読み込んでおきます。サンプルなのでテキストの内容自体は適当です。
import pandas as pd
import ipywidgets as widgets
from IPython.display import display
# サンプルとして適当なテキストの集合を作る
df = pd.DataFrame(
{
"text": [f"ラベル付されるテキストその{i:03d}" for i in range(100)],
"label": None
}
)
print(df.head())
"""
text label
0 ラベル付されるテキストその000 None
1 ラベル付されるテキストその001 None
2 ラベル付されるテキストその002 None
3 ラベル付されるテキストその003 None
4 ラベル付されるテキストその004 None
"""前回の記事ではあまり名前空間のことを考えずにいろいろ書いていましたが、各種ウィジェットがグローバルスコープにあると予期せぬ挙動につながったりもするので、今回はアノテーションのクラスを定義します。
コードはやや長いですがやっていることはシンプルで、渡されたラベルの数だけボタンを作り、データフレーム内のテキストデータを1つ表示し、ボタンが押されたらボタンに対応したlabelのidをデータフレームに書き込んで、また次のテキストを表示するというそれだけです。
そして、全データ処理し終わった時に、ボタンを非活性化する処理も入れています。
今回はサンプルデータが適当で、データ中に何番目のテキストなのか入ってますが、それとは別に i 番目のデータだって表示する機能も入れています。実務では何かしら進捗がわかる機能をつけた方が良いです。どこまで進んだかわからない長いアノテーションは心が折れます。
class annotation():
def __init__(self, df, labels):
self.data = df
self.i = 0 # 何番目のデータをアノテーションしているかのカウンタ
self.buttons = [widgets.Button(description=label) for label in labels]
self.output = widgets.Output()
# ボタンにvalueプロパティを持たせておく
for j in range(len(self.buttons)):
self.buttons[j].value = j
# ボタンにクリック寺の処理を追加
for button in self.buttons:
button.on_click(self.select_label)
# ツールの初期表示実行
self.display_tools()
def display_tools(self):
# ボタンや出力領域の初期表示
self.hbox = widgets.HBox(self.buttons)
self.output.clear_output()
display(self.hbox, self.output)
with self.output:
print(f"{self.i}番目のデータ:\n")
print(self.data.iloc[self.i]["text"])
def select_label(self, button):
# ボタンを押した時の処理
# データフレームにセットされたボタンのvalueを書き込む
self.data.iloc[self.i]["label"] = button.value
# 次のデータに移行
self.i += 1
# 次のデータを表示
self.output.clear_output(True)
# 全データ処理し終わったら完了
if self.i >= len(self.data):
with self.output:
print("完了!")
for button in self.buttons:
button.disabled = True # ボタンを非活性化
return
with self.output:
print(f"{self.i}番目のデータ:\n")
print(self.data.iloc[self.i]["text"])上で作ったクラスは次のように使います。
labels = ["カテゴリー0", "カテゴリー1", "カテゴリー2", "カテゴリー3"]
tool = annotation(df, labels)こうすると次の画像のようにボタンが表示され、クリックするごとにデータフレームに値が書き込まれていきます。

適当のぽちぽちとボタンを押していくと、DataFrameに次のように値が入ります。
print(df.head())
"""
text label
0 ラベル付されるテキストその000 1
1 ラベル付されるテキストその001 2
2 ラベル付されるテキストその002 0
3 ラベル付されるテキストその003 3
4 ラベル付されるテキストその004 0
"""ボタンの配置をもっと整えたり、スキップボタン、戻るボタンの実装などいろいろカスタマイズは考えられますが、一旦これで最低限の役割は果たせると思います。
次にもう一つ、MeCabのユーザー辞書作成の補助ツールを作っておきます。
これは、ボタンを押したらそのボタンに紐づいたラベルが記録されるのではなく、
ドロップダウンで品詞情報を選び、さらに、原形、読み、発音を手入力で入れて、確定ボタンを押したら単語辞書作成用の配列に結果格納するというものです。
今回の記事はお試しなので、追加する単語は適当にピックアップした数個ですが本来はそのリストを作らないといけないので、こちらの記事を参考にしてください。
参考: gensimでフレーズ抽出
また、アウトプットのMeCabユーザー辞書の書式はこちらです。
参考: MeCabでユーザー辞書を作って単語を追加する
ではやってみます。今回も各種パーツを一つのクラスにまとめます。
class create_dictionaly():
def __init__(self, new_words):
self.data = new_words # 辞書に追加する候補のワード
self.i = 0 # カウンタ
self.results = [] # 結果格納用の配列
# 確定とスキップの2種類のボタンを用意する
self.decision_button = widgets.Button(description="確定")
self.decision_button.on_click(self.decision_click)
self.skip_button = widgets.Button(description="スキップ")
self.skip_button.on_click(self.skip_click)
# 品詞の選択機能(サンプルコードなのでIPA辞書の品詞の一部だけ実装)
self.pos_list = [
"名詞,一般,*,*,*,*,*",
"名詞,固有名詞,一般,*,*,*,*",
"名詞,サ変接続,*,*,*,*,*",
"名詞,ナイ形容詞語幹,*,*,*,*,*",
"名詞,形容動詞語幹,*,*,*,*,*",
"名詞,固有名詞,人名,一般,*,*,*",
"名詞,固有名詞,人名,姓,*,*,*",
"名詞,固有名詞,人名,名,*,*,*",
"名詞,固有名詞,組織,*,*,*,*",
]
self.pos_select = widgets.Dropdown(options=self.pos_list)
# 原形, 読み, 発音を設定する項目
self.base = widgets.Text(description="原形: ")
self.reading = widgets.Text(description="読み: ")
self.pronunciation = widgets.Text(description="発音: ")
# 次の単語候補表示場所
self.new_word = widgets.Output()
self.display_tools()
def display_tools(self):
# ボタンや出力領域の初期表示
self.text_hbox = widgets.HBox(
[self.base, self.reading, self.pronunciation])
self.button_hbox = widgets.HBox(
[self.decision_button, self.skip_button])
display(self.new_word, self.text_hbox,
self.pos_select, self.button_hbox)
# 最初の単語を表示しておく
self.next_word()
def next_word(self):
# 次の単語の表示
# 全データ処理し終わったら完了
if self.i >= len(self.data):
self.new_word.clear_output(True)
with self.new_word:
print("完了!")
self.decision_button.disabled = True
self.skip_button.disabled = True
return
self.word = self.data[self.i]
self.new_word.clear_output(True)
with self.new_word:
print(self.word)
self.base.value = self.word
self.reading.value = ""
self.pronunciation.value = ""
self.pos_select.value = self.pos_list[0]
self.i += 1
def decision_click(self, button):
# 確定ボタンクリック
# MeCabユーザー辞書の形式のテキストを生成
result_text = f"{self.word},,,,{self.pos_select.value},"
result_text += f"{self.base.value},{self.reading.value},{self.pronunciation.value}"
# 結果の一覧に格納
self.results.append(result_text)
# 次の単語表示
self.next_word()
def skip_click(self, button):
# スキップボタンクリック
# 次の単語表示
self.next_word()実行は次のコードです。
cd_tool = create_dictionaly(["スマホ", "クラウド", "ガジェット", "インターフェース", "ブログ"])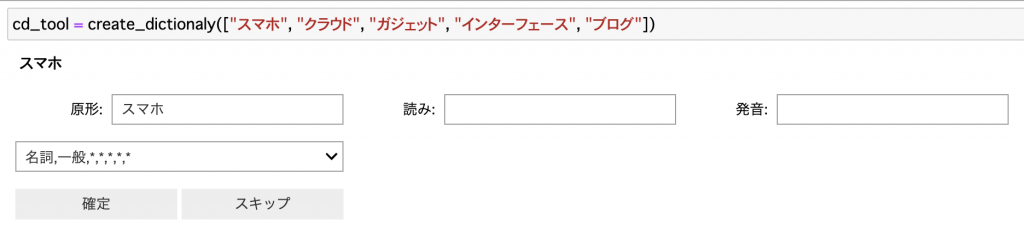
このように、単語候補が出現し、原形(一旦そのままの値で補完)と読み、発音を入力して、品詞を選んで確定を押すと辞書用のデータが記録されていく仕組みになっています。
結果は、cd_tool.result に入ってます。
print("\n".join(cd_tool.results))
"""
スマホ,,,,名詞,一般,*,*,*,*,*,スマホ,スマホ,スマホ
クラウド,,,,名詞,一般,*,*,*,*,*,クラウド,クラウド,クラウド
ガジェット,,,,名詞,一般,*,*,*,*,*,ガジェット,ガジェット,ガジェット
インターフェース,,,,名詞,一般,*,*,*,*,*,インターフェース,インターフェース,インターフェース
ブログ,,,,名詞,一般,*,*,*,*,*,ブログ,ブログ,ブログ
"""これをテキストファイルに書き出せば、ユーザー辞書のseedデータになります。
原形/読み/発音はテキストボックスを一つにまとめて自分でカンマを打つ方が早いかもと思ったり、全体的に配置のデザインがイケて無いなとか思うところはあるのですが、この先徐々に改良していきたいと思います。
以上の二つの例で、ボタンの結果をそのまま記録していくパターン、何かしらのウィジェットでデータを入力してボタンを押して確定するパターンの二つを紹介できたので、ラベル付のタスクであれば、これらの組み合わせで大抵は対応できるのではないでしょうか。