以前の記事で読み込んだ手書き数字文字データ(MINIST)を使って、0~9の数字を判定するモデルを作ってみます。
kerasのサンプルコードもあるのですが、せっかくなので少しだけパラメーターなどを変えてやってみましょう。
最初にライブラリをインポートしてデータを準備します。
前処理として配列の形をConv2Dのinputに合わせるのと、
0〜1への正規化、 ラベルの1hot化を行います。
# ライブラリの読み込み
from keras.datasets import mnist
from keras.utils import to_categorical
from keras.models import Sequential
from keras.layers import Dense, Dropout, Flatten
from keras.layers import Conv2D, MaxPooling2D
from keras.callbacks import EarlyStopping
import matplotlib.pyplot as plt
from sklearn.metrics import classification_report
# データの読み込み
(data_train, target_train), (data_test, target_test) = mnist.load_data()
# Conv2D の inputに合わせて変形
X_train = data_train.reshape(-1, 28, 28, 1)
X_test = data_test.reshape(-1, 28, 28, 1)
# 特徴量を0~1に正規化する
X_train = X_train / 255
X_test = X_test / 255
# ラベルを1 hot 表現に変換
y_train = to_categorical(target_train, 10)
y_test = to_categorical(target_test, 10)
続いてモデルの構築です
# モデルの構築
model = Sequential()
model.add(Conv2D(16, kernel_size=(3, 3),
activation='relu',
input_shape=(28, 28, 1)))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Conv2D(32, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(64, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(10, activation='softmax'))
model.compile(
loss="categorical_crossentropy",
optimizer="adam",
metrics=['accuracy']
)
print(model.summary())
# 以下、出力
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
conv2d_1 (Conv2D) (None, 26, 26, 16) 160
_________________________________________________________________
max_pooling2d_1 (MaxPooling2 (None, 13, 13, 16) 0
_________________________________________________________________
dropout_1 (Dropout) (None, 13, 13, 16) 0
_________________________________________________________________
conv2d_2 (Conv2D) (None, 11, 11, 32) 4640
_________________________________________________________________
max_pooling2d_2 (MaxPooling2 (None, 5, 5, 32) 0
_________________________________________________________________
dropout_2 (Dropout) (None, 5, 5, 32) 0
_________________________________________________________________
flatten_1 (Flatten) (None, 800) 0
_________________________________________________________________
dense_1 (Dense) (None, 64) 51264
_________________________________________________________________
dropout_3 (Dropout) (None, 64) 0
_________________________________________________________________
dense_2 (Dense) (None, 10) 650
=================================================================
Total params: 56,714
Trainable params: 56,714
Non-trainable params: 0
_________________________________________________________________
そして学習です。
# 学習
early_stopping = EarlyStopping(
monitor='val_loss',
min_delta=0.0,
# patience=2,
)
history = model.fit(X_train, y_train,
batch_size=128,
epochs=30,
verbose=2,
validation_data=(X_test, y_test),
callbacks=[early_stopping],
)
# 以下出力
Train on 60000 samples, validate on 10000 samples
Epoch 1/30
- 18s - loss: 0.6387 - acc: 0.7918 - val_loss: 0.1158 - val_acc: 0.9651
Epoch 2/30
- 18s - loss: 0.2342 - acc: 0.9294 - val_loss: 0.0727 - val_acc: 0.9772
Epoch 3/30
- 17s - loss: 0.1827 - acc: 0.9464 - val_loss: 0.0571 - val_acc: 0.9815
Epoch 4/30
- 18s - loss: 0.1541 - acc: 0.9552 - val_loss: 0.0519 - val_acc: 0.9826
Epoch 5/30
- 18s - loss: 0.1359 - acc: 0.9598 - val_loss: 0.0420 - val_acc: 0.9862
Epoch 6/30
- 17s - loss: 0.1260 - acc: 0.9620 - val_loss: 0.0392 - val_acc: 0.9880
Epoch 7/30
- 18s - loss: 0.1157 - acc: 0.9657 - val_loss: 0.0381 - val_acc: 0.9885
Epoch 8/30
- 19s - loss: 0.1106 - acc: 0.9673 - val_loss: 0.0349 - val_acc: 0.9889
Epoch 9/30
- 17s - loss: 0.1035 - acc: 0.9694 - val_loss: 0.0359 - val_acc: 0.9885
学習の進み方をプロットしておきましょう。
# Epoch ごとの正解率と損失関数のプロット
fig = plt.figure(figsize=(12, 12))
ax = fig.add_subplot(2, 1, 1, title="loss")
ax.plot(history.epoch, history.history["loss"], label="train_loss")
ax.plot(history.epoch, history.history["val_loss"], linestyle="-.", label="val_loss")
ax.legend()
ax = fig.add_subplot(2, 1, 2, title="acc")
ax.plot(history.epoch, history.history["acc"], label="train_acc")
ax.plot(history.epoch, history.history["val_acc"], linestyle="-.", label="val_acc")
ax.legend()
plt.show()
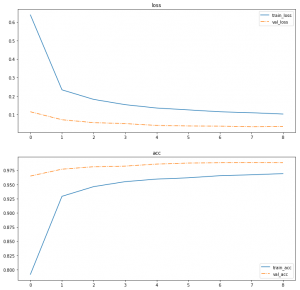
val_acc は結構高い値を出していますが、一応クラスごとの成績も評価しておきましょう。
# 評価
y_predict = model.predict_classes(X_train)
print(classification_report(target_train, y_predict))
# 以下出力
precision recall f1-score support
0 0.99 1.00 0.99 5923
1 0.99 1.00 0.99 6742
2 0.99 0.99 0.99 5958
3 0.99 0.99 0.99 6131
4 0.99 0.99 0.99 5842
5 0.99 0.99 0.99 5421
6 0.99 0.99 0.99 5918
7 0.98 0.99 0.99 6265
8 0.99 0.97 0.98 5851
9 0.98 0.99 0.98 5949
avg / total 0.99 0.99 0.99 60000
ほとんど適当に作ったモデルでしたが、
ほぼほぼ正解できていますね。